
 | Documentation du Job |
| Generated by Talend Open Studio for Data Integration |
| Nom du projet | BDD_Phyto | Date de génération | 3 sept. 2012 14:04:11 |
| Créé par : | alain.benard@nancy.inra.fr | Talend Open Studio VERSION | 5.0.0.r72978 |
| Propriétés | Valeurs |
|---|---|
| Nom | BDD_Phyto |
| Langue | java |
| Description | Base de données Phyto écologie (Floristique - Dendro(s) - Sol) |
| Propriétés | Valeurs |
|---|---|
| Nom | Creation_etude |
| Créé par : | alain.benard@nancy.inra.fr |
| Version | 1.0 |
| Objectif | Création des études et fiches métadonnées associées. |
| Statut | PROD |
| Description | Alimentation de la table des études (t_etude_etu) et création des fiches métadonnées prêtes pour l'importation sous géon |
| Création | 17 juin 2011 16:26:50 |
| Modification | 3 sept. 2012 10:28:29 |
| Paramètres supplémentaires |
| Nom | Valeur |
|---|---|
| COMP_DEFAULT_FILE_DIR | D:/tos/TOS_DI-Win32-r72978-V5.0.0/workspace |
| Exécution multi thread | false |
| tContextLoad implicite | false |
| Stats & Logs |
| Nom | Valeur |
|---|---|
| Utiliser les statistiques (tStatCatcher) | false |
| Utiliser les logs (tLogCatcher) | true |
| Utiliser les volumes (tFlowMeterCatcher) | false |
| Dans la console | true |
| Dans des fichiers | false |
| Dans la base de données | false |
| Capturer les statistiques des composants | false |
| Capturer les erreurs de l'exécutable | true |
| Capturer les erreurs de l'utilisateur | true |
| Capturer les alertes à l'utilisateur | true |
| Contexte :avignon |
| Nom | Prompt | Need Prompt? | Type | Valeur | Source |
|---|---|---|---|---|---|
| dossier_base | dossier_base? | false | id_String | D:/tos/donnees/geonetavignon/global | |
| sous_dossier_tmp | sous_dossier_tmp? | false | id_String | /Integration/tmp | |
| sous_dossier_modeles | sous_dossier_modeles? | false | id_String | /Modeles | |
| sous_dossier_log | sous_dossier_log? | false | id_String | /Integration/log | |
| fichierbdnff | fichierbdnff? | false | id_String | D:/projets/BDD Phyto/codifications/bdnffv5.xlsx | |
| fichiernum_taxo_null | fichiernum_taxo_null? | false | id_String | D:/projets/BDD Phyto/codifications/log/num_taxo_null.csv | |
| fichiernum_taxo_doublon | fichiernum_taxo_doublon? | false | id_String | D:/projets/BDD Phyto/codifications/doublon_numtaxo.csv | |
| fichiergenres | fichiergenres? | false | id_String | D:/projets/BDD Phyto/codifications/Dictionnaire_des_noms_de_genres_utf8.dic | |
| fichiergenre_bddseul | fichiergenre_bddseul? | false | id_String | D:/projets/BDD Phyto/codifications/rejet_genresbddseul.csv | |
| fichiergenre_dicoseul | fichiergenre_dicoseul? | false | id_String | D:/projets/BDD Phyto/codifications/rejet_genresdicoseul.csv | |
| fichierbdnbe | fichierbdnbe? | false | id_String | //mandragore/phyto/bdd/base_phyto/floristique/Liste_des_bryophytes_d'Europe_2008.xls |
| Contexte :Default |
| Nom | Prompt | Need Prompt? | Type | Valeur | Source |
|---|---|---|---|---|---|
| BDD_Encodage | BDD_Encodage? | false | id_String | null | CTX_BDD |
| GEO_Encodage | GEO_Encodage? | false | id_String | null | CTX_BDD |
| dossier_base | dossier_base? | false | id_String | null | CTX_Chemins |
| sous_dossier_log | sous_dossier_log? | false | id_String | null | CTX_Chemins |
| sous_dossier_modeles | sous_dossier_modeles? | false | id_String | null | CTX_Chemins |
| sous_dossier_tmp | sous_dossier_tmp? | false | id_String | null | CTX_Chemins |
| nom_contactMD | nom_contactMD? | false | id_String | null | CTX_Operateur |
| nom_etude | nom_etude? | false | id_String | null | CTX_Operateur |
| num_etude | num_etude? | false | id_Integer | 0 | CTX_Operateur |
| organisation_contactMD | organisation_contactMD? | false | id_String | null | CTX_Operateur |
| DB_Geonetwork_Database | DB_Geonetwork_Database? | false | id_String | db_phyto | DB_Geonetwork |
| DB_Geonetwork_Login | DB_Geonetwork_Login? | false | id_String | admin | DB_Geonetwork |
| DB_Geonetwork_Password | DB_Geonetwork_Password? | false | id_Password | ****** | DB_Geonetwork |
| DB_Geonetwork_Port | DB_Geonetwork_Port? | false | id_String | 5432 | DB_Geonetwork |
| DB_Geonetwork_Schema | DB_Geonetwork_Schema? | false | id_String | public | DB_Geonetwork |
| DB_Geonetwork_Server | DB_Geonetwork_Server? | false | id_String | bdd.nancy.inra.fr | DB_Geonetwork |
| DB_Phyto_Database | DB_Phyto_Database? | false | id_String | ||
| DB_Phyto_Login | DB_Phyto_Login? | false | id_String | ||
| DB_Phyto_Password | DB_Phyto_Password? | true | id_Password | ****** | |
| DB_Phyto_Port | DB_Phyto_Port? | false | id_String | ||
| DB_Phyto_Schema | DB_Phyto_Schema? | false | id_String | ||
| DB_Phyto_Server | DB_Phyto_Server? | false | id_String |
| Contexte :production |
| Nom | Prompt | Need Prompt? | Type | Valeur | Source |
|---|---|---|---|---|---|
| BDD_Encodage | BDD_Encodage? | false | id_String | null | CTX_BDD |
| GEO_Encodage | GEO_Encodage? | false | id_String | null | CTX_BDD |
| dossier_base | dossier_base? | false | id_String | //mandragore/phyto/BDD/base_phyto/floristique | CTX_Chemins |
| sous_dossier_log | sous_dossier_log? | false | id_String | /Integration/log | CTX_Chemins |
| sous_dossier_modeles | sous_dossier_modeles? | false | id_String | /Modeles | CTX_Chemins |
| sous_dossier_tmp | sous_dossier_tmp? | false | id_String | /Integration/tmp | CTX_Chemins |
| nom_contactMD | nom_contactMD? | true | id_String | Alain Benard | CTX_Operateur |
| nom_etude | nom_etude? | false | id_String | null | CTX_Operateur |
| num_etude | num_etude? | false | id_Integer | 0 | CTX_Operateur |
| organisation_contactMD | organisation_contactMD? | true | id_String | INRA | CTX_Operateur |
| DB_Geonetwork_Database | DB_Geonetwork_Database? | false | id_String | DB_Geonetwork | |
| DB_Geonetwork_Login | DB_Geonetwork_Login? | false | id_String | albenard | DB_Geonetwork |
| DB_Geonetwork_Password | DB_Geonetwork_Password? | false | id_Password | ****** | DB_Geonetwork |
| DB_Geonetwork_Port | DB_Geonetwork_Port? | false | id_String | 5432 | DB_Geonetwork |
| DB_Geonetwork_Schema | DB_Geonetwork_Schema? | false | id_String | public | DB_Geonetwork |
| DB_Geonetwork_Server | DB_Geonetwork_Server? | false | id_String | pggeodb.nancy.inra.fr | DB_Geonetwork |
| DB_Phyto_Database | DB_Phyto_Database? | false | id_String | db_phyto | |
| DB_Phyto_Login | DB_Phyto_Login? | false | id_String | albenard | |
| DB_Phyto_Password | DB_Phyto_Password? | true | id_Password | ****** | |
| DB_Phyto_Port | DB_Phyto_Port? | false | id_String | 5432 | |
| DB_Phyto_Schema | DB_Phyto_Schema? | false | id_String | public | |
| DB_Phyto_Server | DB_Phyto_Server? | false | id_String | pggeodb.nancy.inra.fr |
| Contexte :test |
| Nom | Prompt | Need Prompt? | Type | Valeur | Source |
|---|---|---|---|---|---|
| BDD_Encodage | BDD_Encodage? | false | id_String | UTF-8 | CTX_BDD |
| GEO_Encodage | GEO_Encodage? | false | id_String | UTF-8 | CTX_BDD |
| dossier_base | dossier_base? | false | id_String | D:/tos/donnees/bdd_phyto/global | CTX_Chemins |
| sous_dossier_log | sous_dossier_log? | false | id_String | /Integration/log | CTX_Chemins |
| sous_dossier_modeles | sous_dossier_modeles? | false | id_String | /Modeles | CTX_Chemins |
| sous_dossier_tmp | sous_dossier_tmp? | false | id_String | /Integration/tmp | CTX_Chemins |
| nom_contactMD | nom_contactMD? | true | id_String | Alain Benard | CTX_Operateur |
| nom_etude | nom_etude? | false | id_String | null | CTX_Operateur |
| num_etude | num_etude? | false | id_Integer | 0 | CTX_Operateur |
| organisation_contactMD | organisation_contactMD? | true | id_String | INRA | CTX_Operateur |
| DB_Geonetwork_Database | DB_Geonetwork_Database? | false | id_String | db_geonetwork | DB_Geonetwork |
| DB_Geonetwork_Login | DB_Geonetwork_Login? | false | id_String | admin | DB_Geonetwork |
| DB_Geonetwork_Password | DB_Geonetwork_Password? | false | id_Password | ****** | DB_Geonetwork |
| DB_Geonetwork_Port | DB_Geonetwork_Port? | false | id_String | 5432 | DB_Geonetwork |
| DB_Geonetwork_Schema | DB_Geonetwork_Schema? | false | id_String | public | DB_Geonetwork |
| DB_Geonetwork_Server | DB_Geonetwork_Server? | false | id_String | bdd.nancy.inra.fr | DB_Geonetwork |
| DB_Phyto_Database | DB_Phyto_Database? | false | id_String | db_phyto | |
| DB_Phyto_Login | DB_Phyto_Login? | false | id_String | admin | |
| DB_Phyto_Password | DB_Phyto_Password? | true | id_Password | ****** | |
| DB_Phyto_Port | DB_Phyto_Port? | false | id_String | 5432 | |
| DB_Phyto_Schema | DB_Phyto_Schema? | false | id_String | public | |
| DB_Phyto_Server | DB_Phyto_Server? | false | id_String | bdd.nancy.inra.fr |
| Nom du composant | Type de composant |
|---|---|
| tDie_1 | tDie |
| tDie_2 | tDie |
| tDie_3 | tDie |
| tPrejob_1 | tPrejob |
| tRunJob_1 | tRunJob |
| tRunJob_2 | tRunJob |
| tRunJob_3 | tRunJob |
| tRunJob_4 | tRunJob |
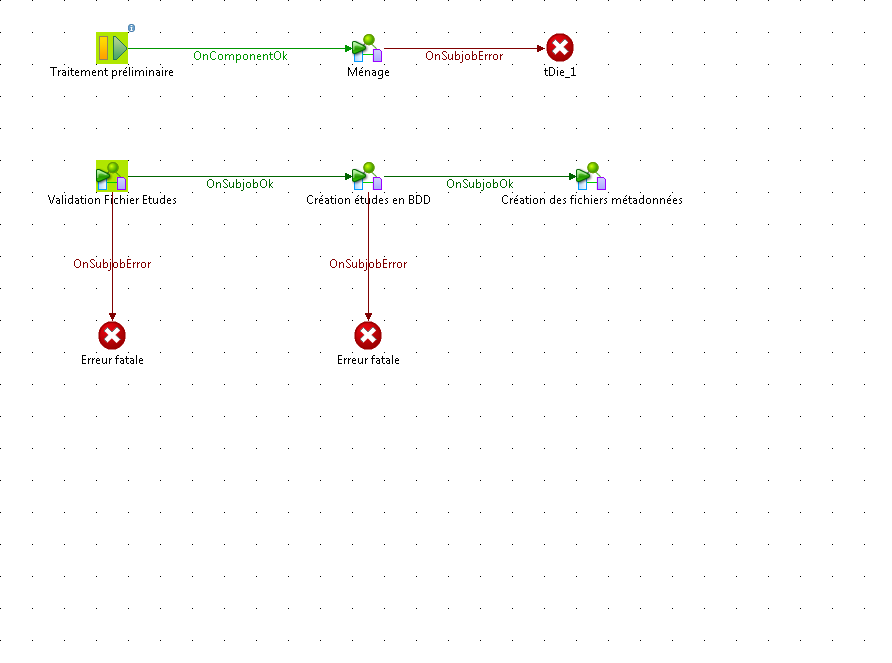
| Composant : tDie |
 | UNIQUE NAME | tDie_1 | INPUT(S) | tRunJob_1 |
| Libellé | __UNIQUE_NAME__ | OUTPUT(S) | none |
| Propriétés | Valeurs |
|---|---|
| Activer | true |
| Statistiques du tStatCatcher | false |
| Message d'arrêt | "Erreur lors de l'exécution du sous-job ménage" |
| Code d'erreur | 4 |
| Priorité | 5 |
| Sortir de la JVM immédiatement | false |
| Afficher les informations | false |
| Commentaire | |
| Utiliser une règle de validation existante | false |
| Column | Clé | Type | Longueur | Précision | Nullable | Commentaire |
|---|
| Composant : tDie |
 | UNIQUE NAME | tDie_2 | INPUT(S) | tRunJob_2 |
| Libellé | Erreur fatale | OUTPUT(S) | none |
| Propriétés | Valeurs |
|---|---|
| Activer | true |
| Statistiques du tStatCatcher | false |
| Message d'arrêt | "Erreur lors de l'exécution du sous-job de validation du fichier des études à créer" |
| Code d'erreur | 4 |
| Priorité | 6 |
| Sortir de la JVM immédiatement | false |
| Afficher les informations | false |
| Commentaire | |
| Utiliser une règle de validation existante | false |
| Column | Clé | Type | Longueur | Précision | Nullable | Commentaire |
|---|
| Composant : tDie |
 | UNIQUE NAME | tDie_3 | INPUT(S) | tRunJob_3 |
| Libellé | Erreur fatale | OUTPUT(S) | none |
| Propriétés | Valeurs |
|---|---|
| Activer | true |
| Statistiques du tStatCatcher | false |
| Message d'arrêt | "Erreur lors de l'exécution du sous-job de création des études en Base de données" |
| Code d'erreur | 4 |
| Priorité | 6 |
| Sortir de la JVM immédiatement | false |
| Afficher les informations | false |
| Commentaire | Ce composant est utilisé dans le cas où aucune étude n'a été inscrite en base de données. |
| Utiliser une règle de validation existante | false |
| Column | Clé | Type | Longueur | Précision | Nullable | Commentaire |
|---|
| Composant : tPrejob |
 | UNIQUE NAME | tPrejob_1 | INPUT(S) | none |
| Libellé | Traitement préliminaire | OUTPUT(S) | tRunJob_1 |
| Propriétés | Valeurs |
|---|---|
| Activer | true |
| Statistiques du tStatCatcher | false |
| Afficher les informations | true |
| Commentaire | Le prétraitement réalise le ménage des précédentes exécutions (log, fichiers temporaires et fiches métadonnées) : Job 'Ménage' |
| Utiliser une règle de validation existante | false |
| Composant : tRunJob |
 | UNIQUE NAME | tRunJob_1 | INPUT(S) | tPrejob_1 |
| Libellé | Ménage | OUTPUT(S) | tDie_1 |
| Propriétés | Valeurs |
|---|---|
| Activer | true |
| Statistiques du tStatCatcher | false |
| Use dynamic job | false |
| Utiliser un processus indépendant pour exécuter le sous-job. | false |
| Arrêt en cas d'erreur du fils | true |
| Transmettre tout le contexte | true |
| Paramètre de contexte | [] |
| Afficher les paramètres | false |
| Afficher les informations | false |
| Commentaire | Voir documentation du sous-job. |
| Utiliser une règle de validation existante | false |
| Column | Clé | Type | Longueur | Précision | Nullable | Commentaire |
|---|
| Composant : tRunJob |
 | UNIQUE NAME | tRunJob_2 | INPUT(S) | none |
| Libellé | Validation Fichier Etudes | OUTPUT(S) | tDie_2, tRunJob_3 |
| Propriétés | Valeurs |
|---|---|
| Activer | true |
| Statistiques du tStatCatcher | false |
| Use dynamic job | false |
| Utiliser un processus indépendant pour exécuter le sous-job. | false |
| Arrêt en cas d'erreur du fils | true |
| Transmettre tout le contexte | true |
| Paramètre de contexte | [] |
| Afficher les paramètres | false |
| Afficher les informations | false |
| Commentaire | Vérifie si le fichier d'entrée présente des anomalies et s'il existe des études pouvant alimenter la suite du traitement : Job. |
| Utiliser une règle de validation existante | false |
| Column | Clé | Type | Longueur | Précision | Nullable | Commentaire |
|---|
| Composant : tRunJob |
 | UNIQUE NAME | tRunJob_3 | INPUT(S) | tRunJob_2 |
| Libellé | Création études en BDD | OUTPUT(S) | tDie_3, tRunJob_4 |
| Propriétés | Valeurs |
|---|---|
| Activer | true |
| Statistiques du tStatCatcher | false |
| Use dynamic job | false |
| Utiliser un processus indépendant pour exécuter le sous-job. | false |
| Arrêt en cas d'erreur du fils | true |
| Transmettre tout le contexte | true |
| Paramètre de contexte | [] |
| Afficher les paramètres | false |
| Afficher les informations | false |
| Commentaire | |
| Utiliser une règle de validation existante | false |
| Column | Clé | Type | Longueur | Précision | Nullable | Commentaire |
|---|
| Composant : tRunJob |
 | UNIQUE NAME | tRunJob_4 | INPUT(S) | tRunJob_3 |
| Libellé | Création des fichiers métadonnées | OUTPUT(S) | none |
| Propriétés | Valeurs |
|---|---|
| Activer | true |
| Statistiques du tStatCatcher | false |
| Use dynamic job | false |
| Utiliser un processus indépendant pour exécuter le sous-job. | false |
| Arrêt en cas d'erreur du fils | true |
| Transmettre tout le contexte | true |
| Paramètre de contexte | [] |
| Afficher les paramètres | false |
| Afficher les informations | false |
| Commentaire | |
| Utiliser une règle de validation existante | false |
| Column | Clé | Type | Longueur | Précision | Nullable | Commentaire |
|---|