
 | Documentation du Job |
| Generated by Talend Open Studio for Data Integration |
| Nom du projet | BDD_Phyto | Date de génération | 3 sept. 2012 14:04:09 |
| Créé par : | alain.benard@nancy.inra.fr | Talend Open Studio VERSION | 5.0.0.r72978 |
| Propriétés | Valeurs |
|---|---|
| Nom | BDD_Phyto |
| Langue | java |
| Description | Base de données Phyto écologie (Floristique - Dendro(s) - Sol) |
| Propriétés | Valeurs |
|---|---|
| Nom | bdnff5 |
| Créé par : | alain.benard@nancy.inra.fr |
| Version | 1.0 |
| Objectif | Intégration des espèces de la BDNFF 5 |
| Statut | PROD |
| Description | ajoutbidon Alimentation de la table des espèces à partir du fichier excel de la base de données BDNFF. Ce tableau présen |
| Création | 6 janv. 2011 14:51:24 |
| Modification | 3 sept. 2012 10:27:02 |
| Paramètres supplémentaires |
| Nom | Valeur |
|---|---|
| COMP_DEFAULT_FILE_DIR | D:/tos/TOS_DI-Win32-r72978-V5.0.0/workspace |
| Exécution multi thread | false |
| tContextLoad implicite | false |
| Stats & Logs |
| Nom | Valeur |
|---|---|
| Utiliser les statistiques (tStatCatcher) | false |
| Utiliser les logs (tLogCatcher) | false |
| Utiliser les volumes (tFlowMeterCatcher) | false |
| Dans la console | false |
| Dans des fichiers | false |
| Dans la base de données | false |
| Capturer les statistiques des composants | false |
| Capturer les erreurs de l'exécutable | true |
| Capturer les erreurs de l'utilisateur | true |
| Capturer les alertes à l'utilisateur | true |
| Contexte :Default |
| Nom | Prompt | Need Prompt? | Type | Valeur | Source |
|---|---|---|---|---|---|
| dossier_base | dossier_base? | false | id_String | null | CTX_Chemins |
| fichierbdnff | fichierbdnff? | false | id_String | null | CTX_Chemins |
| fichiernum_taxo_doublon | fichiernum_taxo_doublon? | false | id_String | null | CTX_Chemins |
| fichiernum_taxo_null | fichiernum_taxo_null? | false | id_String | null | CTX_Chemins |
| sous_dossier_log | sous_dossier_log? | false | id_String | null | CTX_Chemins |
| sous_dossier_modeles | sous_dossier_modeles? | false | id_String | null | CTX_Chemins |
| sous_dossier_tmp | sous_dossier_tmp? | false | id_String | null | CTX_Chemins |
| DB_Phyto_Database | DB_Phyto_Database? | false | id_String | ||
| DB_Phyto_Login | DB_Phyto_Login? | false | id_String | ||
| DB_Phyto_Password | DB_Phyto_Password? | true | id_Password | ****** | |
| DB_Phyto_Port | DB_Phyto_Port? | false | id_String | ||
| DB_Phyto_Schema | DB_Phyto_Schema? | false | id_String | ||
| DB_Phyto_Server | DB_Phyto_Server? | false | id_String | ||
| id_flore_origine | id_flore_origine? | false | id_Integer | Variables |
| Contexte :production |
| Nom | Prompt | Need Prompt? | Type | Valeur | Source |
|---|---|---|---|---|---|
| dossier_base | dossier_base? | false | id_String | //mandragore/phyto/BDD/base_phyto/floristique | CTX_Chemins |
| fichierbdnff | fichierbdnff? | false | id_String | D:/projets/BDD Phyto/codifications/bdnffv5.xlsx | CTX_Chemins |
| fichiernum_taxo_doublon | fichiernum_taxo_doublon? | false | id_String | //mandragore/phyto/bdd/base_phyto/floristique/integration/log/doublon_numtaxo.csv | CTX_Chemins |
| fichiernum_taxo_null | fichiernum_taxo_null? | false | id_String | //mandragore/phyto/bdd/base_phyto/floristique/integration/log/num_taxo_null.csv | CTX_Chemins |
| sous_dossier_log | sous_dossier_log? | false | id_String | /Integration/log | CTX_Chemins |
| sous_dossier_modeles | sous_dossier_modeles? | false | id_String | /Modeles | CTX_Chemins |
| sous_dossier_tmp | sous_dossier_tmp? | false | id_String | /Integration/tmp | CTX_Chemins |
| DB_Phyto_Database | DB_Phyto_Database? | false | id_String | db_phyto | |
| DB_Phyto_Login | DB_Phyto_Login? | false | id_String | albenard | |
| DB_Phyto_Password | DB_Phyto_Password? | true | id_Password | ****** | |
| DB_Phyto_Port | DB_Phyto_Port? | false | id_String | 5432 | |
| DB_Phyto_Schema | DB_Phyto_Schema? | false | id_String | public | |
| DB_Phyto_Server | DB_Phyto_Server? | false | id_String | pggeodb.nancy.inra.fr | |
| id_flore_origine | id_flore_origine? | false | id_Integer | 5 | Variables |
| Contexte :test |
| Nom | Prompt | Need Prompt? | Type | Valeur | Source |
|---|---|---|---|---|---|
| dossier_base | dossier_base? | false | id_String | D:/tos/donnees/bdd_phyto/global | CTX_Chemins |
| fichierbdnff | fichierbdnff? | false | id_String | D:/projets/BDD Phyto/codifications/bdnffv5.xlsx | CTX_Chemins |
| fichiernum_taxo_doublon | fichiernum_taxo_doublon? | false | id_String | D:/projets/BDD Phyto/codifications/doublon_numtaxo.csv | CTX_Chemins |
| fichiernum_taxo_null | fichiernum_taxo_null? | false | id_String | D:/projets/BDD Phyto/codifications/log/num_taxo_null.csv | CTX_Chemins |
| sous_dossier_log | sous_dossier_log? | false | id_String | /Integration/log | CTX_Chemins |
| sous_dossier_modeles | sous_dossier_modeles? | false | id_String | /Modeles | CTX_Chemins |
| sous_dossier_tmp | sous_dossier_tmp? | false | id_String | /Integration/tmp | CTX_Chemins |
| DB_Phyto_Database | DB_Phyto_Database? | false | id_String | db_phyto | |
| DB_Phyto_Login | DB_Phyto_Login? | false | id_String | admin | |
| DB_Phyto_Password | DB_Phyto_Password? | true | id_Password | ****** | |
| DB_Phyto_Port | DB_Phyto_Port? | false | id_String | 5432 | |
| DB_Phyto_Schema | DB_Phyto_Schema? | false | id_String | public | |
| DB_Phyto_Server | DB_Phyto_Server? | false | id_String | bdd.nancy.inra.fr | |
| id_flore_origine | id_flore_origine? | false | id_Integer | 5 | Variables |
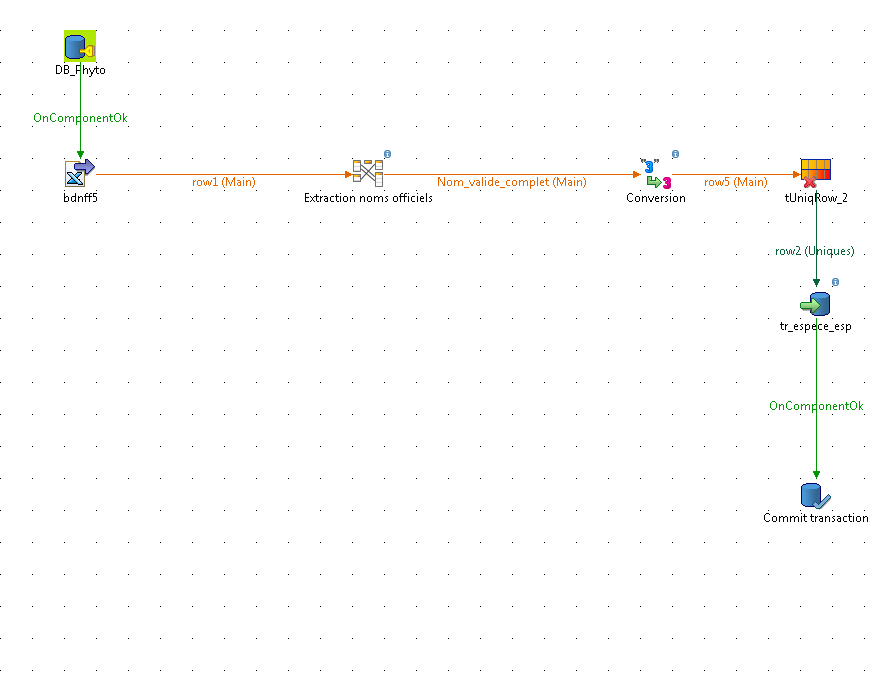
| Nom du composant | Type de composant |
|---|---|
| tConvertType_2 | tConvertType |
| tFileInputExcel_1 | tFileInputExcel |
| tMap_1 | tMap |
| tPostgresqlCommit_1 | tPostgresqlCommit |
| tPostgresqlConnection_1 | tPostgresqlConnection |
| tPostgresqlOutput_1 | tPostgresqlOutput |
| tUniqRow_2 | tUniqRow |
| Composant : tConvertType |
 | UNIQUE NAME | tConvertType_2 | INPUT(S) | tMap_1 |
| Libellé | Conversion | OUTPUT(S) | tUniqRow_2 |
| Propriétés | Valeurs |
|---|---|
| Activer | true |
| Statistiques du tStatCatcher | false |
| !!!IN_SCHEMA.NAME!!! | |
| Conversion automatique | true |
| Définir les valeurs vides comme Null avant de convertir | false |
| Terminer en cas d'erreur | false |
| Afficher les informations | true |
| Commentaire | Ce composant convertit le champ numérique num_taxonomique (fichier BDNFF) en chaîne de caractère pour respecter le type du champ cible de la base de données. |
| Utiliser une règle de validation existante | false |
| Column | Clé | Type | Longueur | Précision | Nullable | Commentaire |
|---|---|---|---|---|---|---|
| nom_espece | false | String | 256 | true | ||
| id_ori | false | Integer | true | |||
| num_taxonomique | false | String | 6 | true |
| Composant : tFileInputExcel |
 | UNIQUE NAME | tFileInputExcel_1 | INPUT(S) | tPostgresqlConnection_1 |
| Libellé | bdnff5 | OUTPUT(S) | tMap_1 |
| Propriétés | Valeurs |
|---|---|
| Activer | true |
| Statistiques du tStatCatcher | false |
| Lire un fichier au format excel2007 (xlsx) | true |
| Nom de fichier/Flux | context.fichierbdnff |
| Toutes les feuilles | true |
| En-tête | 1 |
| Pied-de-page | 0 |
| Limite | |
| Affecte chaque feuille (en-tête et pied de page) | false |
| Première colonne | 1 |
| Dernière colonne | |
| Terminer en cas d'erreur | false |
| Séparateur avancé (pour les nombres) | false |
| Supprimer les espaces en début et en fin de champ dans toutes les colonnes | false |
| Sélectionnez la colonne dans laquelle vous voulez supprimer les espaces entourant les champs | column name="Liste des composants">tPostgresqlConnection_1</column> <column name="Table">"tr_espece_esp"</column> <column name="Action sur la table">NONE</column> <column name="Action sur les données">INSERT</column> <column name="Schéma"></column> <column name="Terminer en cas d'erreur">false</column> <column name="Colonnes supplémentaires">[]</column> <column name="Utiliser les options des champs">false</column> <column name="Activer le mode débogage">false</column> <column name="Supporter des valeurs null dans la clause "WHERE SQL"">false</column> M=false}, {SCHEMA_COLUMN=Aff___Valide__Comb_et_Annee_et_Source, TRIM=false}, {SCHEMA_COLUMN=Aff___egal_synonyme, TRIM=false}, {SCHEMA_COLUMN=Calc___Classif__et_Famille, TRIM=false}, {SCHEMA_COLUMN=Calc___egal_Nom_valide, TRIM=false}, {SCHEMA_COLUMN=calc__Basionyme, TRIM=false}, {SCHEMA_COLUMN=Calc__doute_du_nom_valide, TRIM=false}, {SCHEMA_COLUMN=Calc__Formule_d_hybridation, TRIM=false}, {SCHEMA_COLUMN=Cle_de_tri__Genre, TRIM=false}, {SCHEMA_COLUMN=CombNomen, TRIM=false}, {SCHEMA_COLUMN=D_Annee, TRIM=false}, {SCHEMA_COLUMN=D_Auteur_principal, TRIM=false}, {SCHEMA_COLUMN=D_Basionyme, TRIM=false}, {SCHEMA_COLUMN=D_Doute_nom_valide, TRIM=false}, {SCHEMA_COLUMN=D_Espece, TRIM=false}, {SCHEMA_COLUMN=D_Genre, TRIM=false}] |
| Encodage | "UTF-8" |
| Terminer la lecture sur ligne vide | false |
| Afficher les informations | false |
| Commentaire | |
| Utiliser une règle de validation existante | false |
| Column | Clé | Type | Longueur | Précision | Nullable | Commentaire |
|---|---|---|---|---|---|---|
| Calc___Nom_et_reference_biblio | false | String | 512 | true | ||
| Nom_binomial | false | String | 256 | true | ||
| nom_valide | false | String | 256 | true | ||
| nom_complet | false | String | 256 | true | ||
| num_nomenclatural | true | Integer | 6 | true | ||
| num_taxonomique | false | Integer | 6 | true | ||
| D_Statut | false | Integer | 2 | true | ||
| I_CombNomenNew | false | String | 256 | true | ||
| I_Rang_taxonomique | false | Integer | 6 | true | ||
| D_Num_nom_valide | false | Integer | 6 | true | ||
| Aff___CombNomValide_et_form_Hybr | false | String | 256 | true | ||
| Aff___Subsp_Invalide | false | String | 256 | true | ||
| Aff___Subsp_Valide | false | String | 256 | true | ||
| Aff___Syn___Comb_et_Annee_et_Source | false | String | 256 | true | ||
| Aff___Valide__Comb_et_Annee_et_Source | false | String | 256 | true | ||
| Aff___egal_synonyme | false | String | 256 | true | ||
| Calc___Classif__et_Famille | false | String | 256 | true | ||
| Calc___egal_Nom_valide | false | String | 256 | true | ||
| calc__Basionyme | false | String | 256 | true | ||
| Calc__doute_du_nom_valide | false | String | 128 | true | ||
| Calc__Formule_d_hybridation | false | String | 128 | true | ||
| Cle_de_tri__Genre | false | String | 64 | true | ||
| CombNomen | false | String | 128 | true | ||
| D_Annee | false | String | true | |||
| D_Auteur_principal | false | String | 64 | true | ||
| D_Basionyme | false | String | 8 | true | ||
| D_Doute_nom_valide | false | String | true | |||
| D_Espece | false | String | true | |||
| D_Genre | false | String | true |
| Composant : tPostgresqlCommit |
 | UNIQUE NAME | tPostgresqlCommit_1 | INPUT(S) | tPostgresqlOutput_1 |
| Libellé | Commit transaction | OUTPUT(S) | none |
| Propriétés | Valeurs |
|---|---|
| Activer | true |
| Statistiques du tStatCatcher | false |
| Liste des composants | tPostgresqlConnection_1 |
| Fermer la connexion | true |
| Afficher les informations | false |
| Commentaire | Commit de la transaction d'insertion des espèces. |
| Utiliser une règle de validation existante | false |
| Column | Clé | Type | Longueur | Précision | Nullable | Commentaire |
|---|
| Composant : tPostgresqlConnection |
 | UNIQUE NAME | tPostgresqlConnection_1 | INPUT(S) | none |
| Libellé | DB_Phyto | OUTPUT(S) | tFileInputExcel_1 |
| Propriétés | Valeurs |
|---|---|
| Activer | true |
| Statistiques du tStatCatcher | false |
| Hôte | context.DB_Phyto_Server |
| Port | context.DB_Phyto_Port |
| Base de données | context.DB_Phyto_Database |
| Schéma | context.DB_Phyto_Schema |
| Utilisateur | context.DB_Phyto_Login |
| Mot de passe | context.DB_Phyto_Password |
| Utiliser ou enregistrer une connexion partagée à une base de données | false |
| Commit automatique | false |
| Afficher les informations | false |
| Commentaire | |
| Utiliser une règle de validation existante | false |
| Composant : tPostgresqlOutput |
 | UNIQUE NAME | tPostgresqlOutput_1 | INPUT(S) | tUniqRow_2 |
| Libellé | tr_espece_esp | OUTPUT(S) | tPostgresqlCommit_1 |
| Propriétés | Valeurs |
|---|---|
| Activer | true |
| Statistiques du tStatCatcher | false |
| Utiliser une connexion existante | true |
| Liste des composants | tPostgresqlConnection_1 |
| Table | "tr_espece_esp" |
| Action sur la table | NONE |
| Action sur les données | INSERT |
| Schéma | |
| Terminer en cas d'erreur | false |
| Colonnes supplémentaires | [] |
| Utiliser les options des champs | false |
| Activer le mode débogage | false |
| Supporter des valeurs null dans la clause "WHERE SQL" | false |
| Utiliser la taille des lots | true |
| Taille des lots | 10000 |
| Afficher les informations | true |
| Commentaire | Table des espèces de la base de données |
| Utiliser une règle de validation existante | false |
| Column | Clé | Type | Longueur | Précision | Nullable | Commentaire |
|---|---|---|---|---|---|---|
| nom_espece | false | String | 256 | true | ||
| id_ori | false | Integer | true | |||
| num_taxonomique | false | String | 6 | true |
| Composant : tUniqRow |
 | UNIQUE NAME | tUniqRow_2 | INPUT(S) | tConvertType_2 |
| Libellé | __UNIQUE_NAME__ | OUTPUT(S) | tPostgresqlOutput_1 |
| Propriétés | Valeurs |
|---|---|
| Activer | true |
| Statistiques du tStatCatcher | false |
| Clé unique | [{CASE_SENSITIVE=false, KEY_ATTRIBUTE=false, SCHEMA_COLUMN=nom_espece}, {CASE_SENSITIVE=false, KEY_ATTRIBUTE=false, SCHEMA_COLUMN=id_ori}, {CASE_SENSITIVE=false, KEY_ATTRIBUTE=true, SCHEMA_COLUMN=num_taxonomique}] |
| Seulement une fois chaque clé dupliquée | false |
| Utiliser le disque (convient au traitement d'un grand nombre de lignes) | false |
| Afficher les informations | false |
| Commentaire | |
| Utiliser une règle de validation existante | false |
| Column | Clé | Type | Longueur | Précision | Nullable | Commentaire |
|---|---|---|---|---|---|---|
| nom_espece | false | String | 256 | true | ||
| id_ori | false | Integer | true | |||
| num_taxonomique | false | String | 6 | true |
| Composant : tMap |
 | UNIQUE NAME | tMap_1 | INPUT(S) | tFileInputExcel_1 |
| Libellé | Extraction noms officiels | OUTPUT(S) | tConvertType_2 |